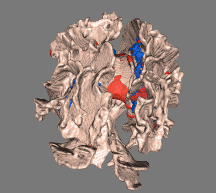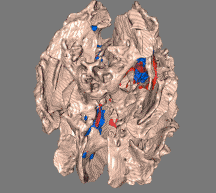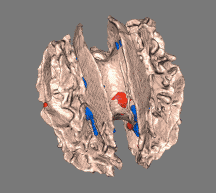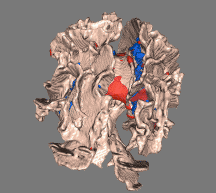
扩散加权磁共振成像(DWI或DW-MRI)是使用特定的MRI序列以及从所得数据生成图像的软件,该软件使用水分子的扩散在MR图像中产生对比度。组织中的分子扩散不是自由的,而是反映了与许多障碍物(例如大分子,纤维和膜)的相互作用。因此,水分子扩散模式可以揭示有关正常或处于患病状态的组织结构的微观细节。弥散张量成像(DTI)是一种特殊的DWI,已被广泛用于绘制大脑中的白质束图。
1. FA数据的三维统计分析
使用磁共振扩散成像通过测量白质束中水的各向异性扩散能够提供有关大脑中解剖学连接性的信息。FA数据的三维统计分析是使用FSL的一部分TBSS(Tract-Based Spatial Statistics,基于空间的空间统计)进行的1。首先,通过拟合张量模型创建FA图像使用FDT提取原始扩散数据,然后使用BET进行脑提取,然后使用非线性配准工具FNIRT将所有受试者的FA数据对齐到一个公共空间中。
接下来,创建平均FA图像并将其细化以创建一个平均FA骨架,该骨架代表该组共有的所有区域的中心,然后将每个对象的对齐FA数据投影到此图像上骨架,然后将所得数据输入到voxelwise跨主题统计信息中。
2. FA参数的计算
分数各向异性(Fractional Anisotropy,FA)是介于0和1之间的标量值,它描述了扩散过程的各向异性程度。零值表示扩散是各向同性的,即在所有方向上都是不受限制的(或同等受限制的)。值为1表示仅沿一个轴发生扩散,而沿所有其他方向完全限制扩散。
定义
扩散椭球完全由扩散张量D表示。FA由特征值($\colorbox{aqua}{$\lambda_1,\lambda_2,\lambda_3$}$)的扩散张量。特征值$\lambda_1$对应的特征向量 $\epsilon_1$给出椭球具有主轴的方向,$\lambda_1$给出该方向上的峰值大小,详细信息见维基百科。
$$ FA=\sqrt{\frac{3}{2}}\frac{\sqrt{(\lambda_1-\bar{\lambda})^2+(\lambda_2-\bar{\lambda})^2+(\lambda_3-\bar{\lambda})^2}}{\sqrt{\lambda_1^2+\lambda_2^2+\lambda_3^2}}, \quad\bar\lambda=(\lambda_1+\lambda_2+\lambda_3)/3 $$
FSL预处理计算FA
首先确保你的数据已经转换为nii格式,常用工具多为dcm2nii(跨平台软件)。主要步骤如下(对每个被试图像都要做):

提取b0像
fslroi sub* nodif 0 1(输入文件为sub*)涡流校正
eddy_correct sub* data.nii.gz 0(输出文件:data.nii.gz)创建大脑面具
bet nodif.nii.gz nodif_brain.nii.gz -f 0.2 -g 0 -m(输出文件:nodif_brain.nii.gz)创建FA图(输出文件:data_FA.nii.gz)
dtifit -k data.nii.gz -o data -m nodif_brain_mask.nii.gz -r bvec -b bval --save_tensor计算其他参数L1、L2、L3(可选)
fslmaths data_L2.nii.gz -add data_L3.nii.gz -div 2 data_L23m
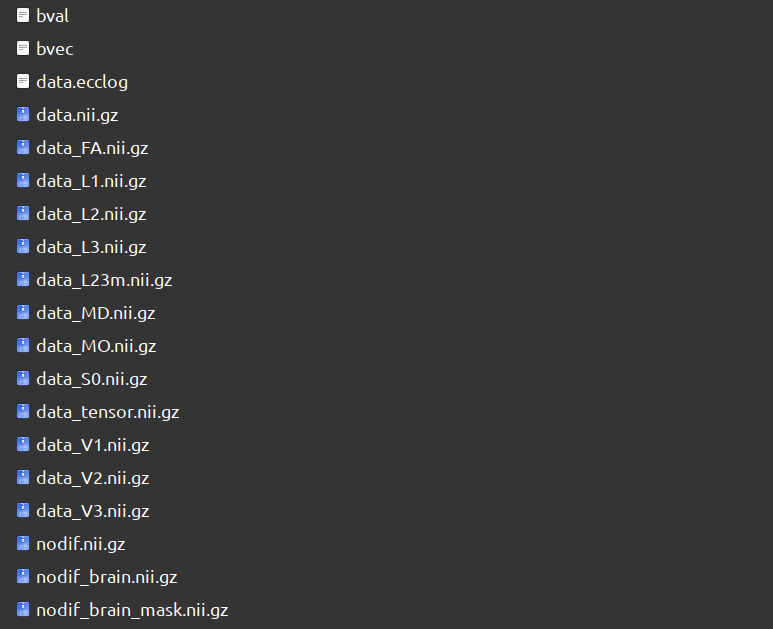
处理完的每个被试文件夹下应该包含以下文件,如图所示。
3. TBSS处理
分数各向异性(FA)是最常从扩散数据中得出的量度之一,它可以量化局部区域结构的定向强度。为了定位与发育,退化和疾病相关的大脑变化,许多影像学研究开始在三维像素统计分析中使用FA图像1。TBSS(基于纤维束的空间统计)旨在提高多主题扩散成像研究分析的敏感性,客观性和可解释性。
运行TBSS步骤
首先,创建新的空的目录文件夹并将所有被试的FA图像复制其中(注意更改被试FA名称)。在当前目录下,运行以下代码:
tbss_1_preproc * .nii.gz
tbss_2_reg -T
tbss_3_postreg -S
tbss_4_prestats 0.2至此,你将得到包含(投影的)骨架化FA数据的4D图像文件(all_FA_skeletonised.nii.gz,在./stats文件夹下)。关于此部分的具体细节可见官网TBSS手册。
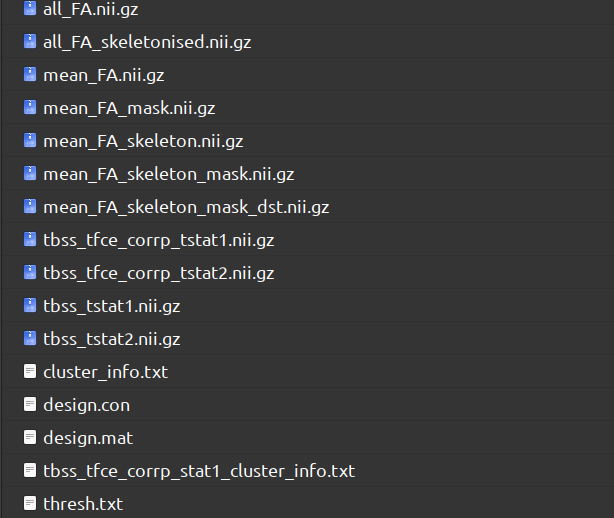
骨架化FA数据的体素统计
有了骨架化FA数据的4D图像文件,就可以进行统计分析(在./stats/路径下操作)。例如,可以探究两组被试之间哪些FA骨架体素显着不同等等。该过程主要包括两个方面:
- 设计矩阵文件(命令行或者GUI)
- 简单的双样本T检验可以用
design_ttest2命令 - 复杂一点就用GUI:命令行
fsl$\to$Misc按钮,具体设计细节根据任务确定,详见手册 - 设计完成后会生成
design.mat及desigan.con文件
- 简单的双样本T检验可以用
- 使用randomise工具进行体素统计和推断
randomise -i all_FA_skeletonised -o tbss -m mean_FA_skeleton_mask -d design.mat -t design.con -n 500 --T2- 原始(无阈值,即未经过校正的)tstat图像分别是
tbss_tstat1和tbss_tstat2,经过充分校正的文件为分别为tbss_tfce_corrp_tstat1和tbss_tfce_corrp_tstat2 cluster命令查看显著性核团:cluster -i tbss_tfce_corrp_tstat1.nii.gz -t 0.95 --mm($p\lt 0.05$),也可以将结果重定向输出到文件
可用如下命令查看统计结果:
# 未校正的统计结果
fslview $FSLDIR/data/standard/MNI152_T1_1mm mean_FA_skeleton -l Green -b 0.2,0.8 tbss_tstat1 -l Red-Yellow -b 3,6 tbss_tstat2 -l Blue-Lightblue -b 3,6
# 校正后的统计结果
fslview $FSLDIR/data/standard/MNI152_T1_1mm mean_FA_skeleton -l Green -b 0.2,0.8 tbss_tfce_corrp_tstat1 -l Red-Yellow -b 3,6 tbss_tfce_corrp_tstat2 -l Blue-Lightblue -b 3,6非FA图像的统计
对于非FA图像的统计其实也很简单,某种程度上来说和FA的一样。你只需要将非FA图像的名称(例如MD等)更改为FA,重复FA数据的统计过程即可。详细说明见手册
$\cdots$ end $\cdots$